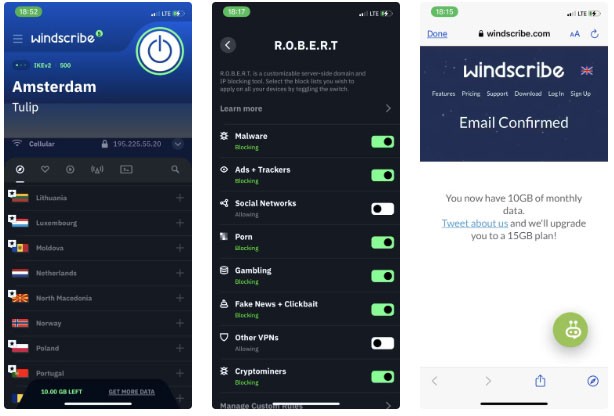
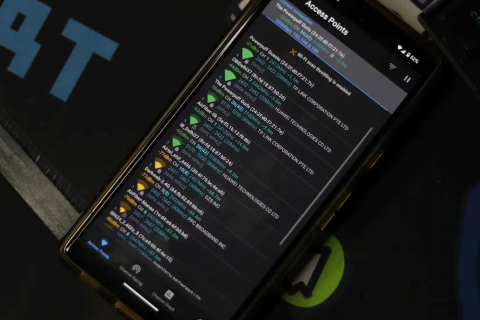
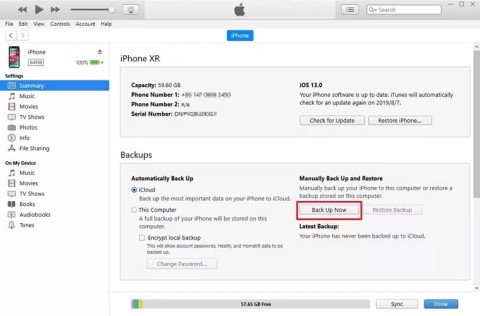
Como recuperar o acesso ao disco rígido, corrigir o erro de não conseguir abrir o disco rígido
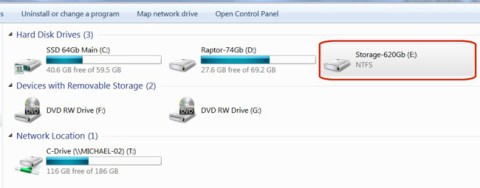
Neste artigo, mostraremos como recuperar o acesso ao seu disco rígido em caso de falha. Vamos acompanhar!
Uma equipe de pesquisa liderada pelo biólogo sintético Xavier Duportet, cofundador da empresa de biotecnologia Eligo Bioscience, projetou com sucesso uma ferramenta de edição genética que pode modificar a população bacteriana na microbiota intestinal de camundongos vivos.
Os resultados experimentais iniciais mostraram que a ferramenta ajudou a modificar o gene alvo em mais de 90% das colônias de Escherichia coli dentro dos intestinos de camundongos vivos. Escherichia coli é um tipo de bactéria que normalmente vive nos intestinos de humanos e animais. Sabe-se que a maioria das cepas de E. coli causa diarreia transitória e leve, ou algumas infecções intestinais graves que levam a doenças mais sérias, como diarreia, dor abdominal e febre.
Vários estudos anteriores usaram o sistema de edição genética CRISPR—Cas para matar bactérias nocivas nos intestinos de camundongos. Mas Duportet e colegas queriam editar os genes das bactérias no microbioma intestinal sem matá-las.
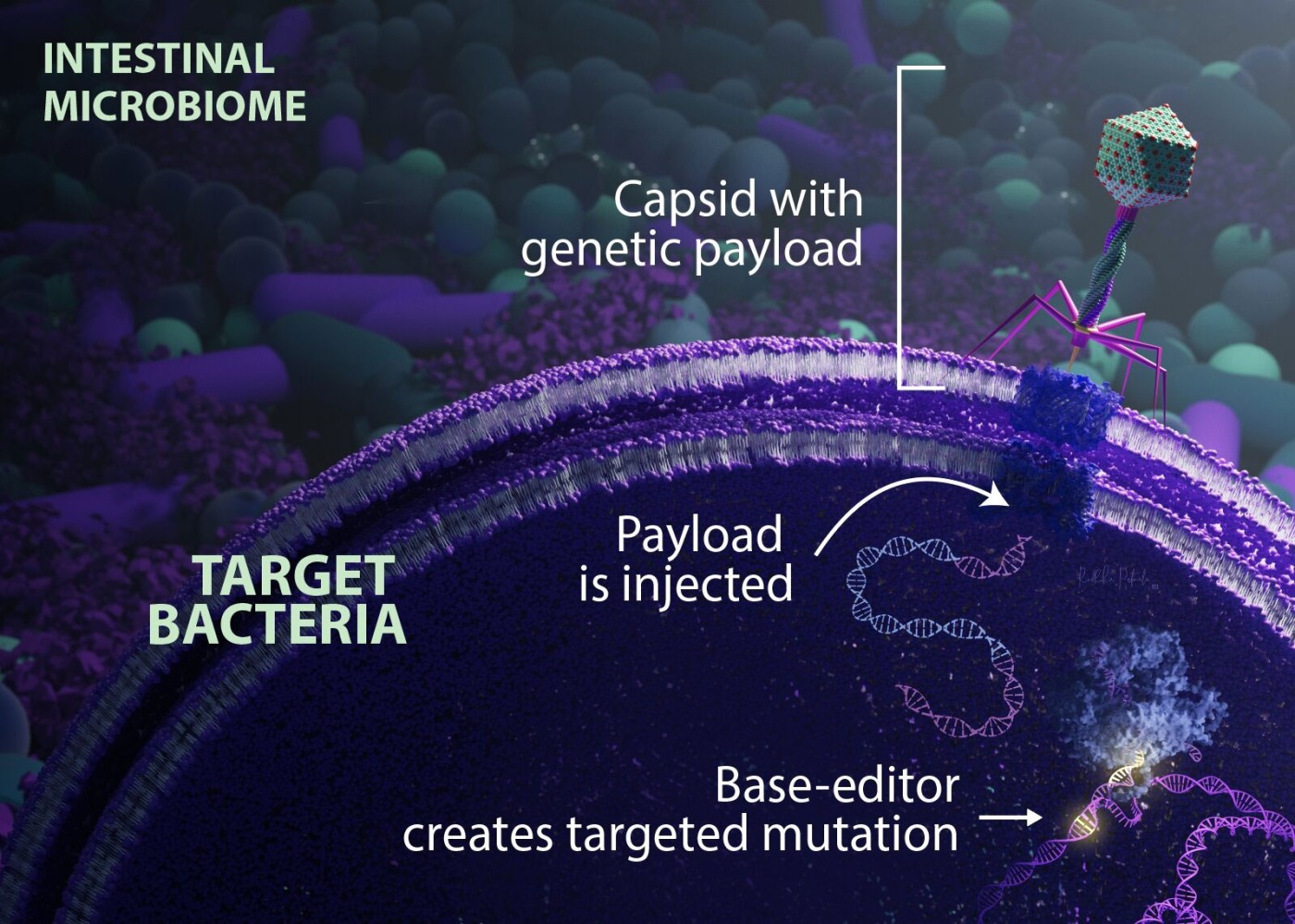
Para fazer isso, os cientistas usaram um método de troca de uma base de nucleotídeo por outra — como converter um A em um G — sem quebrar a fita dupla do DNA. Até o momento, a maioria dos métodos existentes não consegue modificar suficientemente a população bacteriana alvo para ser eficaz. Isso ocorre porque os vetores introduziram apenas receptores-alvo comumente encontrados em bactérias cultivadas em laboratório.
Para superar esse obstáculo, Duportet e colegas projetaram um veículo de entrega que usa componentes de um bacteriófago — um tipo de vírus que infecta bactérias — para transportar vários receptores de E. coli expressos no ambiente intestinal. O vetor carrega uma “ferramenta básica de edição genética” que tem como alvo genes específicos de E. coli. A equipe também aprimorou o sistema para evitar que o material genético fornecido se replique e se espalhe quando estiver dentro da bactéria.
A equipe inseriu a ferramenta básica de edição genética em camundongos e a usou para mudar um A para um G em um gene da E. coli que produz β-lactamase — uma enzima que torna as bactérias resistentes a certos antibióticos. Cerca de oito horas após os animais serem tratados, cerca de 93% das bactérias alvo foram geneticamente editadas.
Os pesquisadores então ajustaram a ferramenta de edição para que ela pudesse modificar o gene E. coli que produz uma proteína que se acredita desempenhar um papel em algumas doenças neurodegenerativas e autoimunes. A porcentagem de bactérias editadas ficou em torno de 70% dentro de três semanas após o tratamento dos camundongos. No laboratório, os cientistas também podem usar a ferramenta para editar cepas de E. coli e Klebsiella pneumoniae que podem causar infecções por pneumonia. Isso sugere que o sistema de edição pode ser adaptado para atingir diferentes cepas e espécies bacterianas.
A conquista representa um “grande salto” no desenvolvimento de ferramentas que podem modificar bactérias diretamente dentro do intestino, abrindo a possibilidade de combater doenças e, ao mesmo tempo, impedir a propagação de DNA prejudicial.
O próximo passo para Duportet e colegas é desenvolver modelos de camundongos com doenças induzidas pelo microbioma para medir se edições genéticas específicas têm efeitos benéficos em sua saúde.
Neste artigo, mostraremos como recuperar o acesso ao seu disco rígido em caso de falha. Vamos acompanhar!
À primeira vista, os AirPods parecem fones de ouvido sem fio comuns. Mas tudo mudou quando alguns recursos pouco conhecidos foram descobertos.
A Apple apresentou o iOS 26 – uma grande atualização com um novo design de vidro fosco, experiências mais inteligentes e melhorias em aplicativos familiares.
Os alunos precisam de um tipo específico de laptop para seus estudos. Ele não deve apenas ser potente o suficiente para um bom desempenho na área de estudo escolhida, mas também compacto e leve o suficiente para ser carregado o dia todo.
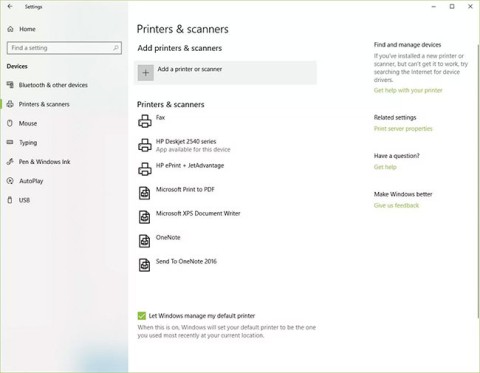
Adicionar uma impressora ao Windows 10 é simples, embora o processo para dispositivos com fio seja diferente do que para dispositivos sem fio.
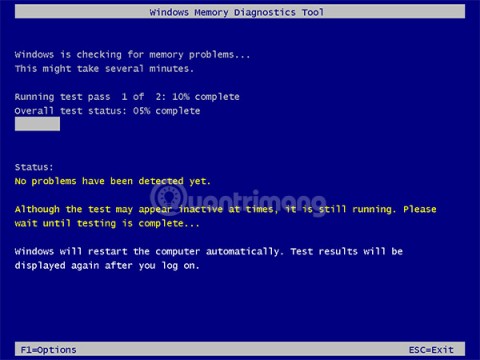
Como você sabe, a RAM é um componente de hardware muito importante em um computador, atuando como memória para processar dados e é o fator que determina a velocidade de um laptop ou PC. No artigo abaixo, o WebTech360 apresentará algumas maneiras de verificar erros de RAM usando software no Windows.
As Smart TVs realmente conquistaram o mundo. Com tantos recursos excelentes e conectividade à Internet, a tecnologia mudou a maneira como assistimos TV.
Geladeiras são eletrodomésticos comuns em residências. As geladeiras geralmente têm 2 compartimentos, o compartimento frio é espaçoso e tem uma luz que acende automaticamente toda vez que o usuário o abre, enquanto o compartimento do freezer é estreito e não tem luz.
As redes Wi-Fi são afetadas por muitos fatores além de roteadores, largura de banda e interferência, mas existem algumas maneiras inteligentes de melhorar sua rede.
Se você quiser voltar para o iOS 16 estável no seu telefone, aqui está o guia básico para desinstalar o iOS 17 e fazer o downgrade do iOS 17 para o 16.
Iogurte é um alimento maravilhoso. É bom comer iogurte todos os dias? Quando você come iogurte todos os dias, como seu corpo muda? Vamos descobrir juntos!
Este artigo discute os tipos de arroz mais nutritivos e como maximizar os benefícios para a saúde de qualquer arroz que você escolher.
Estabelecer um horário de sono e uma rotina para dormir, mudar o despertador e ajustar a dieta são algumas das medidas que podem ajudar você a dormir melhor e acordar na hora certa pela manhã.
Alugue, por favor! Landlord Sim é um jogo de simulação para dispositivos móveis para iOS e Android. Você jogará como proprietário de um complexo de apartamentos e começará a alugar um apartamento com o objetivo de reformar o interior do seu apartamento e deixá-lo pronto para inquilinos.
Obtenha o código do jogo Bathroom Tower Defense Roblox e resgate recompensas emocionantes. Elas ajudarão você a melhorar ou desbloquear torres com maior dano.